Beispiel: Hauptkomponentenanalyse 2
Verwenden Sie die Funktionen Nipals und Nipals2, um komplexe Daten wie die Nah-Infrarot-Spektren von pharmazeutischen Tabletten mit fünf verschiedenen Dosierungen aktiver Inhaltsstoffe zu analysieren (Daten von Bruker Optics, Inc.). Obwohl die tatsächlichen Dosierungen nicht bekannt sind, kann ein Modell erstellt werden, um die einzelnen Dosierungen anhand des Spektrums zu unterscheiden. Das Modell kann bei der Produktion weiterer Tabletten zur Qualitätskontrolle eingesetzt werden.
1. Definieren Sie den folgenden Datensatz
|
|
Dieser Datensatz beschreibt eine Doppelblindstudie eines klinischen Versuchs.
|

Die erste Spalte enthält die Wellendichte (1/Wellenlänge) in cm-1. Es gibt fünf aufeinanderfolgende Spektren jeder Dosierung, welche die restlichen 25 Spalten bilden.
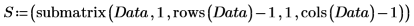
3. Verwenden Sie die Funktionen max und match, um den maximalen Wert und das Spektrum zu ermitteln, das diesen enthält. Da die Datenwerte sehr klein sind, legen Sie TOL auf einen noch kleineren Wert fest.
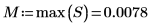
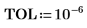
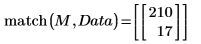
Der maximale Wert befindet sich in Zeile 210 und Spalte 17 der Datenmatrix.
4. Plotten Sie die ersten beiden Spektren jeder Dosierung, also insgesamt 10 Datensätze. Die Datensatzpaare sind die Spalten [1,2], [6,7], [11,12], [16,17] und [21,22] der Matrix Data.
◦ Um einen angemessenen Maßstab für die horizontale Achse zu erhalten, teilen Sie die Wellendichte durch 1000. Da die Spektrumswerte sehr klein sind, multiplizieren wir diese ebenso mit 1000.
◦ Verwenden Sie einen horizontalen Markierer, um den maximalen Spektrumswert anzuzeigen.
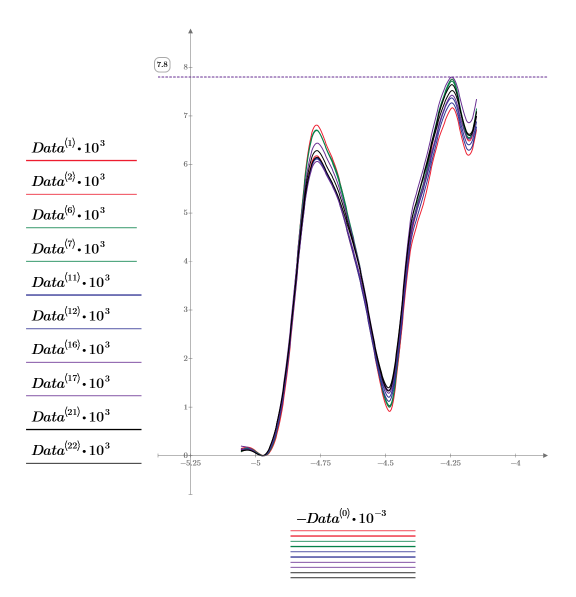
◦ Die Wellendichte wird üblicherweise in absteigender Reihenfolge dargestellt. Data<0> wird deshalb verneint, um die Wellendichte in der richtigen Reihenfolge anzuzeigen.
◦ Kein Teil des Spektrums kann verwendet werden, um eine Dosierung leicht von einer anderen zu unterscheiden: Sie alle haben die gleiche grundlegende Form und ähnliche Absorptionswerte.
◦ Die meisten Daten sind redundant. Jedes Spektrum umfasst 236 Punkte, d.h. 236 gemessene Variablen (Absorptionsvermögen für eine bestimme Wellenlänge von Licht), aber die Variation dieser Punkte ist eindeutig zusammenhängend.
5. Unterteilen Sie die Matrix Data in zwei Datensätze: Wellendichte (Spalte 0) und Spektrum der einzelnen Tabletten (Submatrix S). Um die übliche Konvention einzuhalten, transponieren Sie die Spektren der einzelnen Tabletten, sodass jede Spalte einer unabhängigen Variablen entspricht.
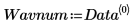
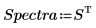
6. Definieren Sie die Anzahl von Hauptkomponenten sowie die Höchstzahl von Iterationen, bevor Sie die Funktion Nipals auf die Daten anwenden. Die Funktion Nipals zentriert die Daten, indem sie das mittlere Spektrum von jeder Zeile subtrahiert.
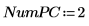
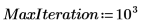
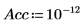
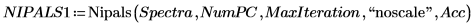
7. Wählen Sie ein zu rekonstruierendes Spektrum.
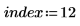
8. Extrahieren Sie die Punkte und die Ladungen aus der Ausgabe der Funktion Nipals.
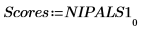
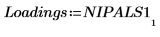
9. Verwenden Sie die Funktion "mean", um das mittlere Spektrum zu berechnen.
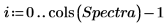
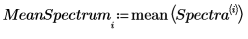
10. Schätzen Sie das ursprüngliche Spektrum, indem Sie die Matrix von Ladungsvektoren mit der Matrix von Punkten multiplizieren und dann das mittlere Spektrum hinzufügen.
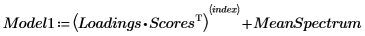
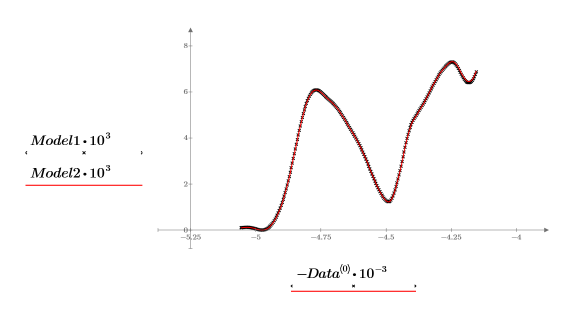
11. Plotten Sie das ursprüngliche und das rekonstruierte Spektrum. Skalieren Sie die horizontale und vertikale Achse, um sinnvolle Werte zu erhalten.
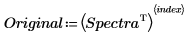
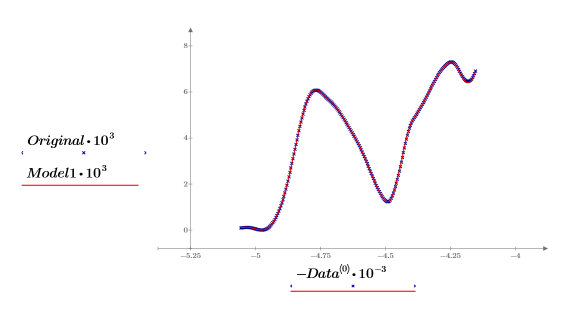
Sämtliche Spektren werden gut dargestellt, wenn nur zwei Hauptkomponenten für die Hauptkomponentenanalyse verwendet werden.
12. Fügen Sie die Punkte in zwei Matrizen ein. Jede Spalte der Matrizen repräsentiert die Punkte für eine der fünf Tablettendosierungen.
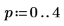
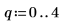
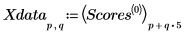
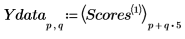
13. Plotten Sie die Punkte des ersten Faktors bezogen auf die Punkte des zweiten Faktors. Jede Dosierung wird in einer anderen Farbe dargestellt.
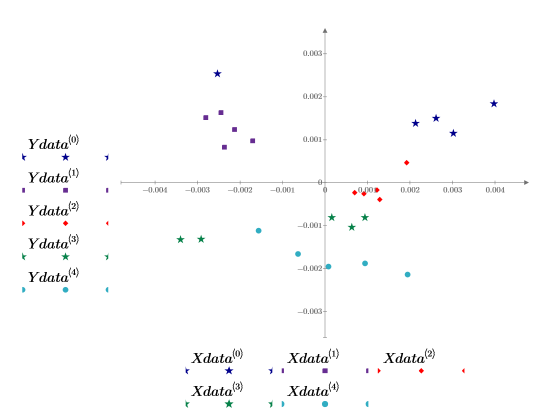
Eine gewisse Gruppierung der Daten ist ersichtlich, aber es ist dennoch schwierig, die einzelnen Dosierungen zu unterscheiden. Das Hinzufügen eines dritten Punktes zum Diagramm könnte helfen.
14. Verwenden Sie die Funktion Nipals2, um dem mit zwei Hauptkomponenten erstellten Modell vier Hauptkomponenten hinzuzufügen.
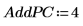
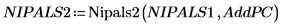
Die Ausgabematrix von NIPALS2 hat die gleiche Form wie die von NIPALS, verfügt entsprechend den zusätzlichen Hauptkomponenten jedoch über zusätzliche Spalten und Zeilen. Die Anzahl der Punkte und Ladungen wurde jetzt auf sechs erhöht.
15. Extrahieren Sie die Ladungen und Punkte aus der Matrix NIPALS2, um ein neues Modell für das gewählte Spektrum zu erstellen.
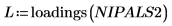
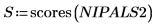
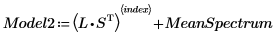
16. Plotten und vergleichen Sie die beiden Modelle für das gewählte Spektrum. Skalieren Sie die horizontale und vertikale Achse, um sinnvolle Werte zu erhalten.
17. Extrahieren Sie die kumulierte Varianz aus NIPALS2.
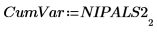
18. Plotten Sie die kumulierte Varianz bezogen auf die Hauptkomponenten.
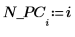
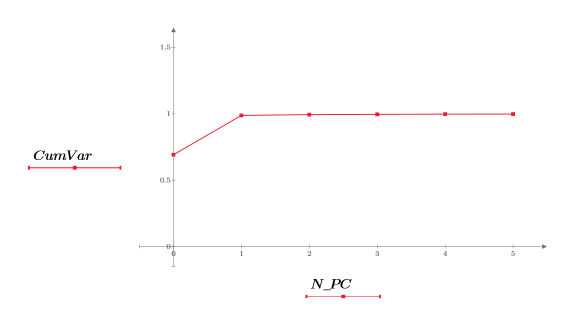
Obwohl die ersten beiden Hauptkomponenten (PC) 99 % der Varianz repräsentieren, ist die dritte Hauptkomponente der Schlüssel zur Gruppierung der Daten nach Dosierung. Durch die Hauptkomponentenanalyse werden die Daten zu den dominantesten, aber nicht den relevantesten Faktoren verdichtet.